研究成果 2019/03/20
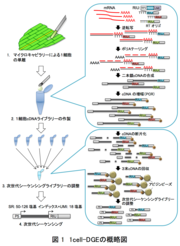
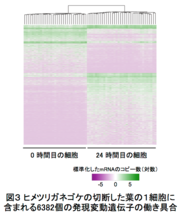
奈良先端科学技術大学院大学(学長:横矢 直和)研究推進機構ヒューマノフィリックイノベーション科学技術研究推進事業の 久保 稔 特任准教授、先端科学技術研究科バイオサイエンス領域の 出村 拓 教授、佐野 亮輔 研究員と、金沢大学、基礎生物学研究所、総合研究大学院大学、広島工科大学、ドイツ フライブルク大学などの国際共同研究グループは、生体内にある個別の細胞について遺伝子の働き具合を調べるための新たな手法の開発に成功しました。マイクロキャピラリー1*という実験器具を用いて生きた組織から1個の細胞を単離する技術と、超高速の解析装置である次世代シーケンサー2*を利用するさいに遺伝子の活性化の指標となるメッセンジャーRNA(mRNA)3*のコピー数を数えるRNA-seq4*という定量法を組み合わせた手法(1cell-DGE)です。さらにこの手法により、モデル植物であるヒメツリガネゴケ5*から植物細胞の初期化、再生に関わる6382個の発現変動遺伝子7*を見出し、葉細胞から幹細胞にリプログラミング6*する過程で幹細胞になる細胞と変化しない細胞の集団の特定に初めて成功しました。
これまでの方法では、個別の細胞について遺伝子の働き具合を調べるときに、細胞をバラバラにすることが前提となっており、それが不可能な生物組織の細胞では、解析ができませんでした。また、細胞をバラバラにすることで隣接する細胞との位置情報が失われるため、他の細胞との関係性を調べることも困難でした。
しかし、今回開発した1cell-DGE法を用いることで、あらゆる生物組織の細胞を分離して、隣接する細胞の位置情報まで利用した1細胞遺伝子発現解析が可能となります。多くの細胞の集まりである組織や器官、個体において、どのように個々の細胞がお互いに協力して、組織、器官や個体を作り上げるのか、また、環境からの刺激に対して組織内の細胞がどのように応答するのかという生き物の形作りや刺激応答の基本的な問題に対して新たな知見を得ることが期待されます。
この研究成果は、平成31年3月15日に英国の国際学術雑誌「Nucleic Acids Research」オンライン版に掲載されました。