2014/08/12
【概要】
奈良先端科学技術大学院大学(奈良先端大 学長:小笠原直毅)・バイオサイエンス研究科・構造生物学研究室の 箱嶋敏雄教授、森智行研究員、平野良憲助教と、東京医科大学・ナノ粒子先端医学応用講座の半田宏特任教授の研究グループ、米国製薬会社Celgene社の 研究グループは国際共同研究を行い、X線結晶構造解析法という手法を使って「サリドマイド」に代表される免疫調整薬(IMiDs、 immunomodulatory drugs)が結合した状態のヒト由来「セレブロン-DDB1」タンパク質の複合体について、三次元立体構造を世界で初めて明らかにした。
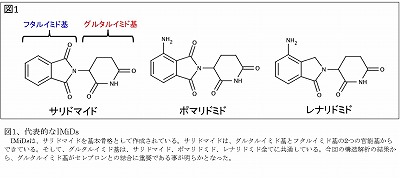
IMiDs は血液のガンである多発性骨髄腫、あるいはハンセン病のらい性結節紅斑をはじめ、さまざまな病気の治療薬として使われはじめている。しかし、IMiDsは サリドマイドを基本にして改良された薬剤であるために(図1)、妊娠初期の女性が服用した場合には、胎児の死産や奇形を起こしてしまう重篤な副作用ももち 合わせている。
IMiDsが生体内のどのような分子と結びついて薬理作用や副作用を発揮するのかは、半世紀以上の間、研究され続けてきた が、不明であった。そこで、2010年に半田宏特任教授らによってサリドマイドと結合するタンパク質として「セレブロン」が同定されたことから急速に研究 の進展が見られるようになった。IMiDsの作用機序は、セレブロンに結合して細胞内の遺伝子の発現を変化させることにある。その結果、ガン細胞の増殖を 抑制することや奇形を引き起こす性質(催奇性)が発揮されることが明らかになってきていた。それでもなお、IMiDsがセレブロンとにどのように結合して いるか、という最も基本的な「IMiDsとセレブロンの結合様式の謎」は残されたままであった。
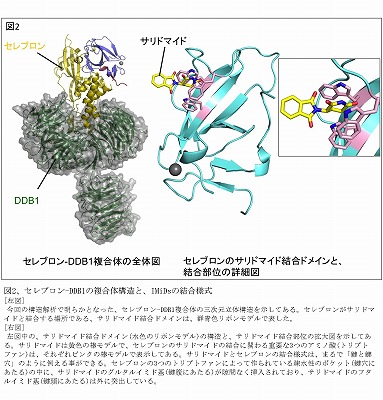
今回の研究成果から、IMiDsとセレブ ロンの結合様式は、セレブロンの表面にある疎水性(水を嫌う性質)のポケット(穴のような空間)にIMiDsが結合している事が明らかになった(図2)。 この三次元構造の解析の結果は、医学、薬学の分野では、非常に価値のある成果であるといえる。今後、立体構造の情報を利用することで、IMiDsの薬理作 用の効果をもっと高めたり、さらには、副作用を取り除いたりするためのIMiDsの改良、開発を「三次元構造を眺めながら極めて効率よく」できるようにな るので、新薬の開発が加速することになるだろう。また、分子生物学、細胞生物学といった基礎研究の分野においても、セレブロンの機能を明らかにしていく上 で、有用な情報も提供できる。
この成果は平成26年8月10日(日)付けのイギリスNature Structural & Molecular Biology誌に掲載される(プレス解禁日時:日本時間 平成26年8月11日 (月)午前2時)。
【解説】
[サリドマイド史]
1950 年代後半から1960年代前半にかけて、サリドマイドは世界30カ国以上で睡眠薬として処方されていた。しかし、副作用として妊娠初期の女性が服用した場 合、死産や、新生児に奇形が生じる事が明らかになり、サリドマイドは処方を制限され、市場から消え去ることになる。しかしながら、その後の研究で、サリド マイドはハンセン氏病や、血液の癌である多発性骨髄腫といった病気の治療に有効である事が明らかとなったことから、厳重な管理のもと使用されはじめ、今で は、より薬理効果の高いIMiDsが開発されるに至っている(図1)。
[研究背景]
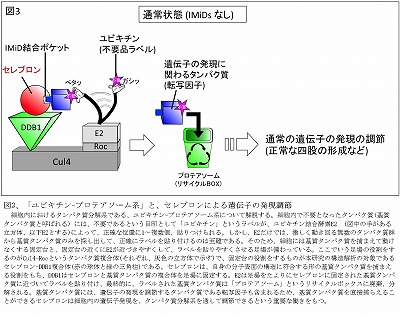
タンパク質は、生命現象を運営、維持するうえ で、極めて重要な働きをもつ。細胞内では、遺伝子発現の最終生産物として、様々な種類のタンパク質が、適切な時に作られて、機能を発揮した後、役割を終え て廃棄、分解される。本研究での構造解析された、セレブロン-DDB1複合体は、細胞内でのタンパク質の廃棄方法の一種である「ユビキチン-プロテアソー ム系」で働くタンパク質である(図3)。
IMiDsの標的タンパク質であるセレブロンは、自身の分子表面の形とぴったり符合できるものを 「基質タンパク質(細胞に不必要な、廃棄するタンパク質)」として捕らえて、タンパク質分解系に送り込む。この基質タンパク質には、細胞の遺伝子の発現を 調節するタンパク質である「転写因子」も含まれている。そのために、セレブロンはタンパク質分解系を通して、細胞の遺伝子の発現を調節する極めて重要な機 能をもっている(図3)。
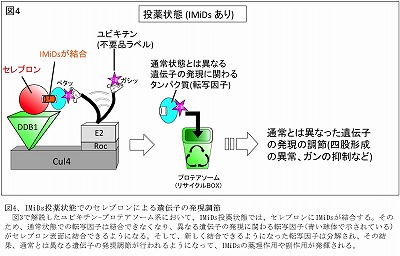
ここに、サリドマイドなどのIMiDsが投薬されると、IMiDsはセレブロンに直接結合して、セレブロンに結合できる転写因子の種類をすり変えて、タンパク質分解系に送る。その結果、細胞内の遺伝子の発現が変化して、薬理効果や副作用を発揮する (図4)。
IMiDs がどのようにセレブロンに結合しているのか、また、どのように基質タンパク質を認識して補えるのか?といった謎は解かれないままであった。IMiDsがど のようにセレブロンに結合しているのかが詳細に分かれば、IMiDsの薬理作用の効果を高めたり、または、副作用を軽減、取り除いたりするための改良、開 発を効率よく進めることができるようになる。また、どのように基質タンパク質を認識して補えるのか?といった、セレブロンの機能を明らかにしていくため の、重要な手がかりが得られると期待されていた。
[研究成果と今後の展開]
本研究では、IMiDsが結合した状態でのセレブロン -DDB1の複合体立体構造を明らかにした。また、セレブロンのIMiDs結合ドメイン(領域)とIMiDsとの複合体の高分解能の立体構造も決定して、 相互作用の詳細を明らかにした。立体構造解析によると、サリドマイドをはじめとするIMiDsは、グルタルイミド基がセレブロンのC末端側に位置する領域 (ジンクフィンガードメイン)の表面にある、3つのトリプトファン残基から作られる疎水性の穴に取り囲まれるようにして結合していた(図2)。
興 味深いことに、サリドマイドを構成するフタルイミド基はセレブロンとの相互作用に寄与せず、セレブロンの外側に突出する形で突き出ている。このことから、 サリドマイドが結合したセレブロンの分子表面は、大きく「付き出された」形になる。このフタルイミド基の突出は、セレブロンが通常の状態とは異なり、新た に別の基質タンパク質を結合させるための形状変化をあたえていると考えられる。実際、サリドマイドよりも薬理作用が強いとされるIMiDsであるポマリド ミドやレナリドミドという物質は、フタルイミド基の部分に改良点があり(図1)、新たに結合する転写因子との結合が強化されているのではないかと推測され る。今回の研究では、IMiDsがセレブロンの表面に結合することによって与える形状変化が、セレブロンと基質タンパク質との結合に充分に影響を変えうる 変化をもたらしていることが明らかとなった。
本研究成果から、セレブロンとIMiDsの結合様式が原子レベルで明らかとなったため、立体構造を基本として、非常に効率的にIMiDsの改良、開発ができるようになる。これは、医学、薬学の分野では、大変意義のある結果であると言える。
今後、今回の研究成果をもとに、さらにセレブロンと基質タンパク質の複合体構造解析や、IMiDs結合状態でのセレブロンと基質タンパク質の複合体解析がなされ、より本質的なセレブロンのタンパク質分解系での機能や、IMiDsの作用機序の解明につながっていくだろう。
[用語解説]
X 線結晶構造解析法:結晶にX線を照射して起こる回折現象を用いて、結晶中の分子の三次元構造を決定する方法。複雑な構造をもつタンパク質や、タンパク質と タンパク質の複合体も、結晶にすることができれば三次元立体構造を原子レベルで決定できる。強力なX線源が必要とされるので、兵庫県にある大型放射光施設 「SPring-8」といった、シンクロトロンでの放射X線が用いられる。
転写因子:遺伝子の本体であるDNAに直接結合して、DNAの遺伝情報のコピーであるRNAの合成を調節する機能をもつタンパク質。RNAにコピーされた情報は、最終的にリボソームと言われるタンパク質合成装置渡され、情報に基づいたタンパク質が合成される。
ジンクフィンガードメイン:タンパク質の機能単位であるドメインの中に、亜鉛(ジンクZinc)が結合したドメイン。本研究で構造解析されたセレブロンには、4残基のシステインが亜鉛の結合に関わっており、サリドマイド結合ドメインの折りたたみに関与している。
【関連リンク】
・論文タイトル
Structure of the human Cereblon-DDB1-lenalidomide complex reveals basis for responsiveness to thalidomide analogs
・論文は以下に掲載されております。
http://dx.doi.org/10.1038/nsmb.2874
・以下は論文の書誌情報です。
Philip P Chamberlain, Antonia Lopez-Girona,Karen Miller, Gilles Carmel, Barbra Pagarigan, Barbara Chie-Leon, Emily Rychak, Laura G Corral, Yan J Ren, Maria Wang, Mariko Riley, Silvia L Delker, Takumi Ito, Hideki Ando, Tomoyuki Mori, Yoshinori Hirano, Hiroshi Handa, Toshio Hakoshima, Thomas O Daniel & Brian E Cathers
Nature Structural & Molecular Biology 21, 803-809 (2014)